on
[PT-BR] Construindo Pipelines de Aprendizagem de Máquina com DVC
Aprenda os princípios para construir pipelines de Aprendizagem de Máquina usando o DVC
Quando os projetos de Aprendizagem de Máquina começam a evoluir quase sempre a gente começa a perder o controle de “que script faz tal coisa” ou “que dados eu usei quando rodei esse experimento?”
O DVC é um sistema de controle de versão por linha de comando para projetos de Aprendizagem de Máquina. Uma das funcionalidades dele é servir como um git para versionar dados, mas a ferramenta vai muito além disso, permitindo que a gente construa e reproduza pipelies de Aprendizagem de Máquina.
Neste artigo você vai aprender como construir pipelines reproduzíveis usando o DVC. O código final pode ser encontrado aqui no GitHub.
 for this [cool picture](https://unsplash.com/photos/9AxFJaNySB8)! ;)](https://cdn-images-1.medium.com/max/2400/1*rwVTHTw7QzNRj8f7HyqtiA.jpeg)
O que iremos construir
Vamos treinar um modelo para classificar o conjunto de dados 20newsgroups. O conjunto contém posts sobre 20 tópicos e vamos selecionar dois destes para tópicos treinar o modelo. Este é o script principal para treinar e avaliar o modelo:
from sklearn.datasets import fetch_20newsgroups
from sklearn.feature_extraction.text import TfidfVectorizer
from sklearn.naive_bayes import MultinomialNB
from sklearn.metrics import precision_recall_curve, auc
# -- Importar os Dados --
categories = ["comp.graphics","sci.space"]
newsgroups_train = fetch_20newsgroups(subset='train', categories=categories)
newsgroups_test = fetch_20newsgroups(subset='test', categories=categories)
newsgroups_all = fetch_20newsgroups(subset='all', categories=categories)
# -- Gerar as Features --
vectorizer = TfidfVectorizer()
vectorizer.fit(newsgroups_all.data)
X = vectorizer.transform(newsgroups_train.data)
# -- Treinar o modelo --
clf = MultinomialNB(alpha=0.1)
clf.fit(X,newsgroups_train.target)
# -- Avaliar o Modelo
X_predict = vectorizer.transform(newsgroups_test.data)
y_pred_scores = clf.predict_proba(X_predict)
y_true = newsgroups_test.target
precision, recall, _ = precision_recall_curve(y_true, y_pred_scores[:, -1])
auc = auc(recall, precision)
print(auc)
Um bom workflow para construir um código limpo e fácil de manter é sempre melhorar a qualidade do código ao longo do desenvolvimento. Tudo bem criar código para experimentar e ver se algo funciona (como feito no código acima). Depois de rodar e ver que tudo está funcionando corretamente podemos melhorar o código e tornar ele mais fácil de manter. Vamos fazer isso usando o DVC.
💡 O princípio para construir pipelines com o DVC
Se você olhar o código do main.py mais de perto pode ver que o script se divide naquela famosa “receita de bolo” de Aprendizagem de Máquina:
1 — Importar os dados
2 — Gerar as features
3 — Treinar o modelo
4 — Avaliar o modelo
Com base dessas etapas, estes são os princípios para construir pipelines com o DVC:
-
Escrever um script para cada uma destas etapas
-
Armazenar os parâmetros que cada script usa em um arquivo
.yaml -
Especificar os arquivos que cada script depende para rodar
-
Especificar os arquivos que cada script gera
Agora vamos instalar o DVC e ver como implementar estes princípios na prática.
🔨 Instalando o DVC
Neste artigo foi utilizado o Linux e o DVC foi instalado como uma biblioteca Python. Para seguir usando o Windows não é muito diferente, você só precisa ter o Python3, pip e Git instalados na sua máquina.
No passo-a-passo você vai criar um ambiente virtual para o projeto (é uma boa prática fazer isso para qualquer projeto em Python). O DVC funciona com um repositório Git, então você vai iniciar um repositório antes de inicializar o projeto com o DVC. Os comandos para fazer tudo isso são:
$ mkdir dvc_tutorial
$ cd dvc_tutorial
$ python3 -m venv .env
$ source .env/bin/activate
(.env)$ pip3 install dvc
(.env)$ git init
(.env)$ dvc init
E agora vamos usar o DVC para implementar cada etapa da pipeline.
📍 1 — Importar os dados
O script usa o método fetch_20newsgroups() do Scikit-learn para carregar os dados na memória, mas o objetivo é salvar os dados em um arquivo para usá-los como dependência para a próxima etapa da pipeline. Então neste primeiro script vamos importar os dados e salvá-los em um arquivo csv.
Com o DVC a gente precisa dar nome a cada estágio, então vamos chamar este estágio de prepare.
De início eu estava usando 3 categorias ['comp.graphics', 'sci.space', 'rec.sport.basecball'], mas ao chegar na etapa de avaliar eu vi que seria mais simples se eu utilizasse apenas duas categorias. Então eu tive que importaar o conjunto de dados novamente, especificando a nova lista de categorias.
Isso me mostrou que é uma boa ideia usar categories como parametro para o script de importar os dados. O DVC usa um arquivo params.yaml com os parâmetros default, então vamos criar um arquivo .yaml e definir as categorias lá:
# arquivo params.yaml
prepare:
categories:
- comp.graphics
- sci.space
prepare é o nome do estágio, categories é o nome do parâmetro e para criar uma lista em yaml usamos um - antes de cada item.
Queremos salvar os dados na pasta data/prepared. Este é o arquivo prepare.py:
# src/prepare.py
import os
import yaml
from sklearn.datasets import fetch_20newsgroups
import pandas as pd
# read params
params = yaml.safe_load(open('params.yaml'))['prepare']
categories = params['categories']
# create folder to save file
data_path = os.path.join('data', 'prepared')
os.makedirs(data_path, exist_ok=True)
#fetch data
newsgroups_train = fetch_20newsgroups(subset='train', categories=categories)
newsgroups_test = fetch_20newsgroups(subset='test', categories=categories)
def newsgroups_to_csv(split_name, newsgroups):
df = pd.DataFrame([newsgroups.data, newsgroups.target.tolist()]).T
df.columns = ['text', 'target']
df_target_names = pd.DataFrame(newsgroups.target_names)
df_target_names.columns = ['target_name']
out = pd.merge(df, df_target_names, left_on='target', right_index=True)
out.to_csv(os.path.join(data_path, split_name+".csv"))
# save data to file
newsgroups_to_csv("train", newsgroups_train)
newsgroups_to_csv("test", newsgroups_test)
Para executar este estágio da pipeline a única dependências é o próprio script, e os dados vão ser salvos na pasta data/prepared. Agora você tem todos os componentes para construir o primeiro estágio da pipeline:
- Escrever o script:
prepare.py - Salvar os parâmetros:
categoriesdentro deparams.yaml - Especificar os arquivos necessários para o script rodar:
prepared.py - Especificar os arquivos que o script gera: a pasta
data/prepared
Para manter o projeto organizar você irá salvar os scripts dentro de uma pasta chamada src. A pasta do seu projeto deve estar assim:
├── params.yaml
└── src
└── prepare.py
Agora é hora de construir este estágio com o DVC.
⏺ dvc run — Construindo Estágios da Pipeline com o DVC
O DVC salva os estágios da pipeline em um arquivo dvc.yaml e um arquivo dvc.lock (para uso interno da biblioteca). Depois de ter criado o script, para criar o estágio da pipeline você utiliza o comando dvc run no terminal. Estas são as principais opções do comando:
-n <stage>: especificar o nome do estágio
-p [<path>:]<params_list>**: especificar os parâmetros que o script precisa para rodar
-d <path>**: especificar os arquivos ou diretórios que o script precisa para rodar
-o <path>: especificar os arquivos ou pastas que o script gera
Após definir estas opções você precisa adicionar o comando do terminal para rodar o script. No nosso caso o comando será python3 scr/prepare.py. Para rodar primeiro você precisa instalar as bibliotecas (scikit, pandas e pyyaml) e depois chamar o dvc run para criar o estágio da pipeline.
(.env)$ pip install pyyaml scikit-learn pandas
(.env)$ dvc run -n prepare -p prepare.categories -d src/prepare.py -o data/prepared python3 src/prepare.py
Depois de rodar o comando acima a pasta do seu projeto deve estar assim:
├── data
│ └── prepared
│ ├── test.csv
│ └── train.csv
├── dvc.lock
├── dvc.yaml
├── params.yaml
└── src
└── prepare.py
E este é o conteúdo de dvc.yaml, gerado automaticamente pelo DVC:
stages:
prepare:
cmd: python3 src/prepare.py
deps:
- src/prepare.py
params:
- prepare.categories
outs:
- data/prepared
Então as etapas até aqui foram: criar um script para importar os dados, definir os parâmetros no arquivo params.yaml e por fim rodar o comando dvc run para registrar este estágio com o DVC e poder reproduzí-lo depois.
🔬 dvc dag — Visualizando a Pipeline
O comando dvc dag mostra os estágios da pipeline. Nossa pipeline só tem um estágio por enquanto:
(.env)$ dvc dag
+---------+
| prepare |
+---------+
~
~
/tmp/tmpixsrsfo0 (END)
Para sair da visualização basta aperta q no teclado.
⏯ dvc repro — Reproduzindo as Pipelines
O comando dvc repro reproduz as pipelines de forma completa ou parcial através da execução dos comandos definidos para cada estágio.
A bibliteca salva em um cache os artefatos relevantes e faz uma busca recursiva nos estágios para determinar quais foram modificados. Se um estágio foi modificado o comando correspondente a ele é executado.
No momento não modificamos nada ainda, então vamos rodar a pipeline novamente para ver o que acontece:
(.env)$ dvc repro
>>> Stage 'prepare' didnt change, skipping
Data and pipelines are up to date.
E se você mudar o nome de uma categoria?
# Arquivo params.yaml
prepare:
categories:
- comp.graphics
- rec.sport.baseball # antes era 'sci.space'
E rodar o comando dvc run novamente?
(.env)$ dvc repro
>>> Running stage 'prepare' with command:
python3 src/prepare.py
Updating lock file 'dvc.lock'
O estágio prepare foi executado novamente porque ao definir o estágio nós adicionamos o parâmetro -p prepare.categories como parâmetro deste estágio. O DVC viu que este parâmetro foi modificado e rodou o estágio novamente. O arquivo dvc.yaml ainda é o mesmo, mas se você olhar o arquivo dvc.lock vai ver que lá os parâmetros mudaram.
Estes são os comandos básicos para criar, rodar e visualizar os estágios da pipeline:
- dvc run
- dvc dag
- dvc repro
Agora é hora de criar o segundo estágio da pipeline, gerando as features para o modelo.
📍 2 — Gerando as Features
O nome deste estágio será featurize e vamos usar o TfidfVectorizer do Scikit-learn para salvar a matrix em um arquivo pickle dentro da pasta data/features. Este estágio depende do script e da pasta data/prepared, então os parâmetros para criá-lo são:
- Escrever o script: featurize.py
- Salvar os parâmetros: (este estágio não precisa de parâmetros)
- Especificar os arquivos necessários para o script rodar:
prepared.pye a pastadata/prepared - Especificar os arquivos que o script gera: a pasta
data/features
Este é o código final do script featurize.py:
# src/featurize.py
import sys
import os
import yaml
import pandas as pd
from sklearn.feature_extraction.text import TfidfVectorizer
import pickle
# ler os parâmetros da linha de comando
if len(sys.argv) != 3:
sys.stderr.write('Arguments error. Usage:\n')
sys.stderr.write(
'\tpython featurize.py data-dir-path features-dir-path\n'
)
sys.exit(1)
data_path = sys.argv[1]
features_path = sys.argv[2]
os.makedirs(features_path, exist_ok=True)
train_input_file = os.path.join(data_path, 'train.csv')
test_input_file = os.path.join(data_path, 'test.csv')
# ler os dados do csv
df_train = pd.read_csv(train_input_file)
df_test = pd.read_csv(test_input_file)
def extract_column(column, df_path):
df = get_df(df_path)
corpus = df[[column]]
return corpus
def get_train_and_test_corpus(df_1, df_2):
corpus_train = df_1["text"]
corpus_test = df_2["text"]
return corpus_train.append(corpus_test)
def append_labels_and_save_pkl(df, tfidf_matrix, filename):
output_file = os.path.join(features_path, filename)
target = df[["target"]]
output = pd.concat([pd.DataFrame(tfidf_matrix.toarray()), target], axis=1)
with open(output_file, 'wb') as f:
pickle.dump(output, f)
vectorizer = TfidfVectorizer()
# precisamos treinar o vectorizer do conjunto train e do conjunto test
corpus = get_train_and_test_corpus(df_train, df_test)
vectorizer.fit(corpus)
# transformar os dados
train_matrix = vectorizer.transform(df_train["text"])
test_matrix = vectorizer.transform(df_test["text"])
# salvar os dados e um pickle (com a coluna das labels)
append_labels_and_save_pkl(df_train, train_matrix, 'train.pkl')
append_labels_and_save_pkl(df_test, test_matrix, 'test.pkl')
Salve este arquivo featurize.py dentro da pasta /src e finalmente crie o estágio usando o comando dvc run novamente:
(.env)$ dvc run -n featurize -d src/featurize.py -d data/prepared -o data/features python3 src/featurize.py data/prepared data/features
Você pode rodar o comando dvc dag novamente para ver o novo estágio adicionado à pipeline.
📍 3 — Treinando o Modelo
Neste estágio você vai treinar o modelo e salvá-lo em um aquivo pickle. Neste exemplo vamos usar o Naive Bayes classifier e o único parâmetro que vamos definir é o alpha. Esta é a receita para gerar este estágio:
- Escrever o script: train.py
- Salvar os parâmetros: o parâmetro
alphadentro deparams.yaml - Especificar os arquivos necessários para o script rodar:
train.pye a pastadata/features - Especificar os arquivos que o script gera: o arquivo
model.pkl
Primeiro vamos adicionar o parâmetro alpha no params.yaml:
# Arquivo params.yaml
prepare:
categories:
- comp.graphics
- sci.space
train:
alpha: 0.1
E por fim salvar o script train.py dentro da pasta /src:
# src/train.py
import sys
import os
import yaml
from sklearn.naive_bayes import MultinomialNB
import pickle
# ler os parâmetros da linha de comando
if len(sys.argv) != 3:
sys.stderr.write('Arguments error. Usage:\n')
sys.stderr.write(
'\tpython3 train.py features-dir-path model-filename\n'
)
sys.exit(1)
features_path = sys.argv[1]
model_filename = sys.argv[2]
# ler os parâmetros da pipeline
params = yaml.safe_load(open('params.yaml'))['train']
alpha = params['alpha']
# carregar as features de treinamento
features_train_pkl = os.path.join(features_path, 'train.pkl')
with open(features_train_pkl, 'rb') as f:
train_data = pickle.load(f)
X = train_data.iloc[:,:-1]
y = train_data.iloc[:,-1]
# treinar o modelo
clf = MultinomialNB(alpha=alpha)
clf.fit(X, y)
# salvar o modelo
with open(model_filename, 'wb') as f:
pickle.dump(clf, f)
Agora você já pode criar este estágio:
(.env)$ dvc run -n train -p train.alpha -d src/train.py -d data/features -o model.pkl python3 src/train.py data/features model.pkl
📍 4 — Avaliando o Modelo
Neste estágio você vai conhecer dois novos parâmetros do comando dvc run: --metrics e --plots:
*-m
Neste exemplo vamos utilizar a métrica Area Under the Curve (AUC) e computar os pares de scores precision-recall com vários thresholds diferentes. Para isso vamos criar um script chamado evaluate.py que criará um arquivo scores.json com o AUC score e outro arquivo plots.json com os pares precision/recall/threshold que serão usados para plotar o gráfico:
# src/evaluate.py
import sys
import os
from sklearn.metrics import precision_recall_curve, auc
import pickle
import json
# ler os parâmetros da linha de comando
if len(sys.argv) != 5:
sys.stderr.write('Arguments error. Usage:\n')
sys.stderr.write(
'\tpython3 evaluate.py model-filename features-dir-path scores-filename\
plots-filename\n'
)
sys.exit(1)
model_filename = sys.argv[1]
features_path = sys.argv[2]
test_features_file = os.path.join(os.path.join(features_path, 'test.pkl'))
scores_file = sys.argv[3]
plots_file = sys.argv[4]
# carregar as features
with open(test_features_file, 'rb') as f:
test_features = pickle.load(f)
X_test = test_features.iloc[:,:-1]
y_test = test_features.iloc[:,-1]
# carregar o modelo
with open(model_filename, 'rb') as f:
model = pickle.load(f)
# fazer as predições
predictions_by_class = model.predict_proba(X_test)
predictions = predictions_by_class[:,-1]
# gerar os scores
precision, recall, thresholds = precision_recall_curve(y_test, predictions)
auc = auc(recall, precision)
# salvar os scores
with open(scores_file, 'w') as f:
json.dump({'auc': auc}, f)
# salvar os dados
with open(plots_file, 'w') as f:
proc_dict = {'proc': [{
'precision': p,
'recall': r,
'threshold': t
} for p, r, t in zip(precision, recall, thresholds)
]}
json.dump(proc_dict, f)
Com relação aos arquivos de métricas e de gráficos temos duas opções:
-
Deixar o DVC gerenciar os arquivos
-
Gerenciar os arquivos usando o Git
Como neste tutorial não vimos como usar o DVC para gerenciar arquivos vamos usar a segunda opção e gerenciar os arquivos usando o Git. Para isso vamos usar os comandos --metrics-no-cache e --plots-no-cache. Esta é a receita que vamos usar para isso:
- Escrever o script:
evaluate.py - Salvar os parâmetros: (este estágio não precisa de parâmetros)
- Especificar os arquivos necessários para o script rodar:
evaluate.py,model.pklanddata/features - Especificar os arquivos que o script gera: (nenhum)
- (NOVO) Especificar os arquivos para as métricas e os gráficos:
scores.jsoneplots.json
Este é o comando para gerar este estágio:
(.env)$ dvc run -n evaluate -d src/evaluate.py -d model.pkl -d data/features --metrics-no-cache scores.json --plots-no-cache plots.json python3 src/evaluate.py model.pkl data/features scores.json plots.json
🏋🏿 dvc metrics — Comparando métricas com o DVC
O comando dvc metrics permite que você mostre e compare métricas. O comando dvc metrics show imprime o valor da métrica e o comando dvc metrics diff mostra a diferença entre dois valores, o atual e o do último commit. Vamos ver tudo isso na prática:
O comando dvc metrics show mostra o score atual:
(.env)$ **dvc metrics show**
scores.json:
auc: 0.9993366236676577
Vamos ver os parâmetros que estão sendo usandos atualmente:
# file params.yaml
prepare:
categories:
- comp.graphics
- rec.sport.baseball
train:
alpha: 0.1
O comando dvc metrics diff calcula a diferença entre o último commit e o estado atual do repositório, então para ver a diferença vamos comitar os arquivos:
(.env)$ git add src/ params.yaml dvc.yaml dvc.lock scores.json plots.json
(.env)$ git commit -m "exp: alpha=0.1"
Hora de mudar o parâmetro alpha para ver como isso afeta o modelo:
# file params.yaml
prepare:
categories:
- comp.graphics
- rec.sport.baseball
train:
alpha: 0.9
E rodar o experimento novamente:
(.env)$ dvc repro
Podemos ver a diferença entre parâmetros com o comando dvc params diff:
(.env)$ dvc params diff
Path Param Old New
params.yaml train.alpha 0.1 0.9
E finalmente podemos ver como os scores foram alterados com o comando dvc metrics diff:
(.env)$ dvc metrics diff
Path Metric Value Change
scores.json auc 0.99869 -0.00064
📊 dvc plots — Visualizando e Comparando as Métricas
O comando dvc plots gera os gráficos em arquivos HTML usando o Vega-Lite. Para plotar a curva de precision-recall nós vamos colocar precision no eixo y e recall no eixo x. Este é o comando para plotar o gráfico:
(.env)$ dvc plots show -y precision -x recall plots.json
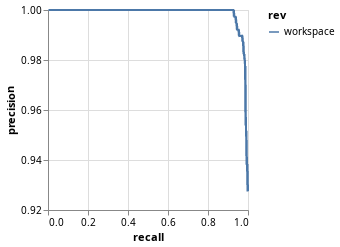
Também é possível plotar a diferença entre os scores para os valores de alpha = 0.1 e alpha = 0.9:
(.env)$ dvc plots diff --targets plots.json -y precision
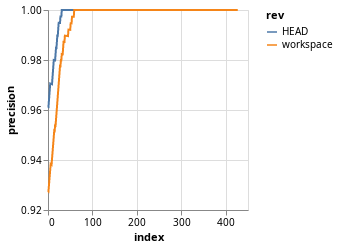
E chegamos ao fim da tour. Você pode ver mais gráficos e configurações diferentes aqui: https://dvc.org/doc/command-reference/plots.
O código final do tutorial pode ser encontrado aqui.
Finalizando
Com o DVC gerenciar os projetos de Aprendizagem de Máquina se torna uma tarefa bem mais fácil. A chave para tornar as pipelines reproduzíveis é criar um script em Python para cada etapa da pipeline e especificar os parâmetros, os arquivos que o script precisa e os arquivos que o script gera. Feito isso você pode utilizar os comandos dvc run e dvc repro para reproduzir a pipeline.
O artigo ficou no gerenciamento de pipelines, mas o DVC também oferece outras features como controle de versão para os dados, colaboração e deployment.
Obrigada pela leitura! 😁